目次
導入
こんにちは。InsightEdgeのデータサイエンティストの小柳です。
本記事ではデータ分析の強い味方、MCMCサンプラーの実装を見てみようと思います。 今回取り上げるのは私が普段使っているPyMCというPython用のモジュールです。
実装を読む動機はいくつかあります。教科書で読むHMCの方法は美しいですが、自分で実装しろと言われるとどうすればよいかすぐにはわかりません。そのような不思議なものがどうやって実際に作られているのかはとても気になります。他にも、PyMCに限りませんがサンプリング前にエラーが起きたときに対処ができるようになりたいとか、わずか数行でサンプリングが動く便利さはどうやって実装されているのか解き明かしたいとか、 特殊な使い方をするときにはどこをどう触ればよいのか知りたいとか、ご利益はたくさんあります。また、PyMCはあまり日本語の文献が無いのでこの界隈を盛り上げたい、そんな動機もあります。
PyMCとは
おそらくこの記事を読むような方には不要かと思いますが、軽くPyMCについての紹介をします。
PyMCとは簡単に言えばPython向けのベイズ推定用のサンプリングモジュールです。確率的プログラミング言語(PPL)とも呼ばれます。Python向けに限らず同等なものはいくつか存在しています。 Pyro, NumPyro, TensorFlowProbability, Stan(PyStan), WinBUGS, JAGSあたりでしょう。どれもMCMCサンプリングと変分ベイズ(変分推論、ADVI)、プラスアルファで確率的最適化ができるはずです。
PyMCの特徴は開発が盛んなこと、そして使用者が多いことです。以下はGoogle Trendsで調べた上記のPython向けPPLの人気です。
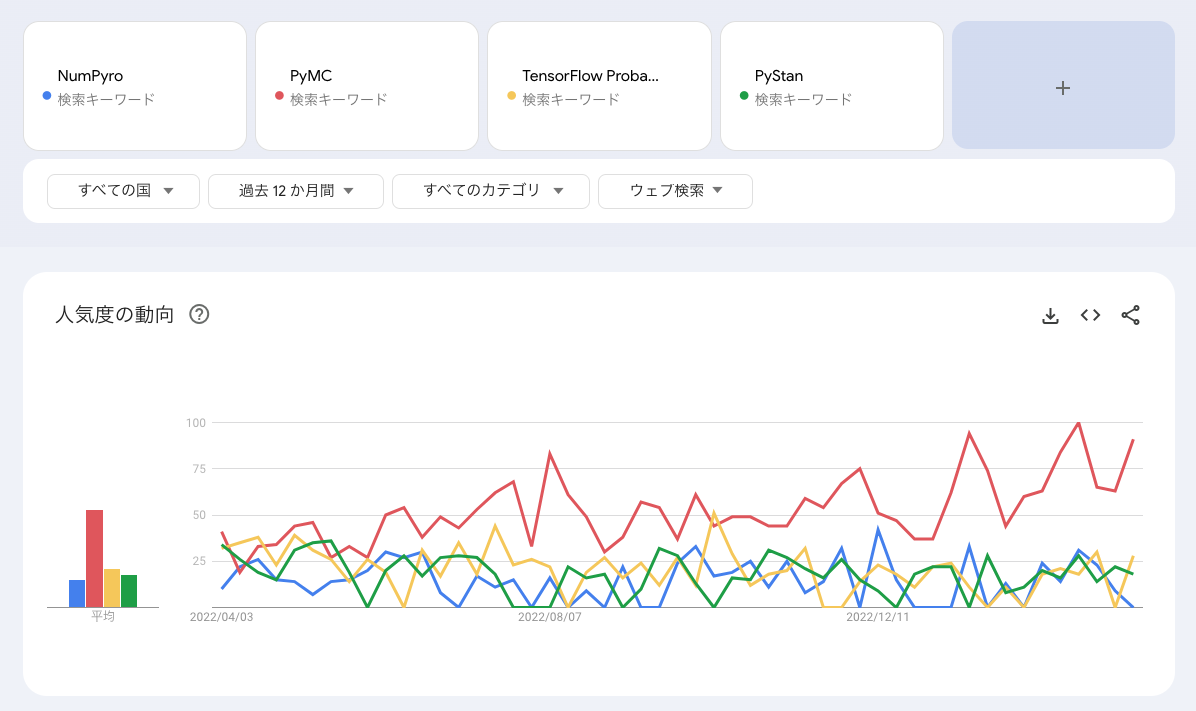
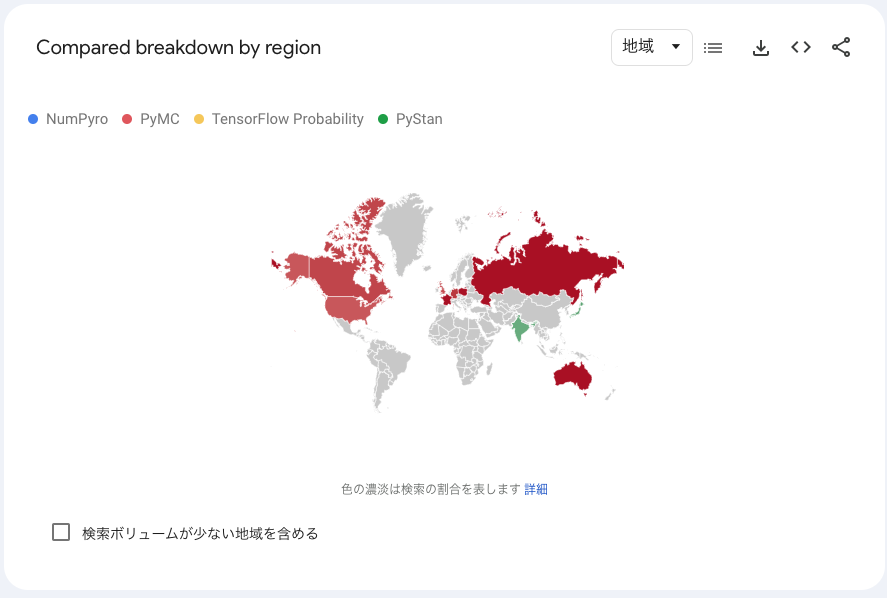
また、国別でいうと、日本ではPyStanがトップ、PyMCとNumPyroが同程度で2位という感じですが、その他のほぼ全ての国ではPyMCが主流といった趣です。
PyMCの最近の動向
2017年にver3系がリリースされたあたりからPyMCはPython向けのPPLの中でもメジャーなものであり続けています。ですが、近年(2022年あたり)の動向をキャッチアップしているものは少ない印象です。PyMCに関することをGoogle検索するとモジュールインポートの際に
import pymc3
と書いている記事が多いのですが、これはそのver3系を使ったものです。最新のものをインポートする際にはpymcでOKです。
このver3のときまで、PyMCはTheanoというモジュールをバックエンドとして使っていましたが、Theanoの開発が2018年で終わってしまったようで[1]別のモジュールを使う方向に進んできました。当初はTensorFlowProbabilityを代替として開発を進めていましたが、その方針も結局2020に破棄[2]。ついにTheanoの後継としてAesaraをそしてそのさらに後ろでJAX等を使う方向になったようです。
紆余曲折ありましたが、ついに、2022年にver4.0がリリースされました[3]。その当時に書かれたイラストが以下です。
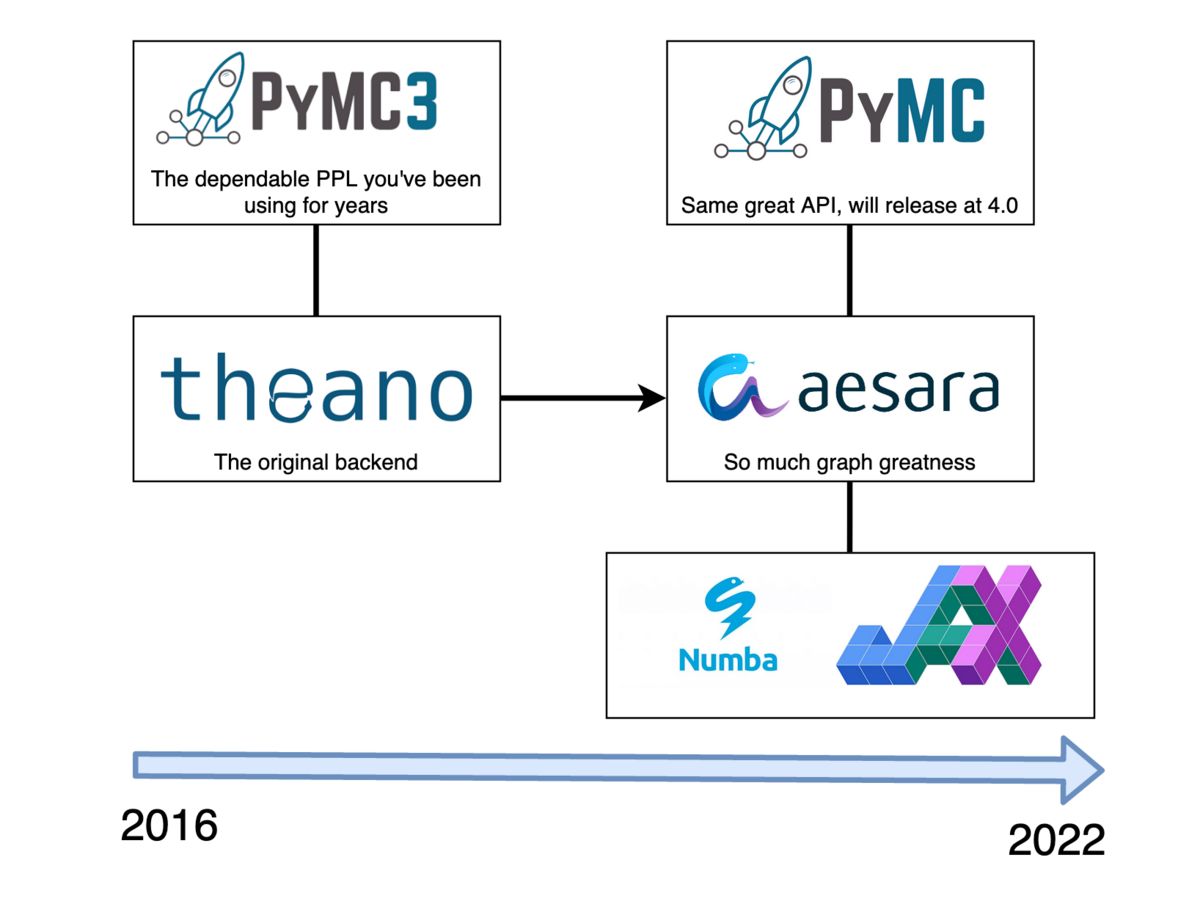
ですがver4.0はとても短命でした。開発目標の違いなどから、今はAesaraからフォークしたPyTensorを使う仕様になるに伴いver5系となり、現在も開発が進められています。
コードリーディングの方針とスコープ
今回はpymc v5.1.0を読んでいこうと思います。 また、読む範囲としてはPyMCが担当している範囲とします。従って、PyTensorやJAXが担当している範囲はスコープ外です。次回以降でやるかもしれませんが… 全てを理解しようとすると膨大になってしまうので、関係が薄い部分は大胆に削っていくことにします。 また、私のバックグラウンドはPythonを使ったデータ分析はできるけれどモジュール開発等まではできない人間です。なのでデータ分析では使わないようなPythonの使い方に重点を置いて読んでいくことにします。 方針としては、以下のような単純なモデルの挙動を追っていくことにします。
import pymc as pm
trials = 10; successes = 5
with pm.Model() as coin_flip_model:
p = pm.Beta("p", alpha=1, beta=1)
obs = pm.Binomial("obs", p=p, n=trials,
observed=successes,
)
idata = pm.sample()
二項分布の事前分布にベータ分布をおいたモデルになります。非常に単純です。共役事前分布なんだからMCMCせず手計算で瞬殺だろと言いたくなるようなモデルですね。 変数名から推測するに10回投げたら5回表がでたようなコインの表がでる確率の事後分布を求めるような問題です。 PyMCのWebサイトのInteractive Demoから引用しています。
メインコンテンツ
Modelクラスとインスタンス化
まずはmodel.py内のModelクラスの挙動から確認していきましょう。
with文
モデル作成時にwith文が使われています。with文の詳細はPythonの公式ドキュメント[5]に譲るのですが、端的に言えばModelクラスの__enter__()メソッドがwith文の冒頭で実行され、同__exit__()メソッドがwith文の最後に実行されます。従って、まずはModelクラスの__new__(), __init__(), __enter__(), __exit__()メソッドを確認していきます。が、その前に分かりづらいのがModelクラス定義時に指定されているmetaclassです。
メタクラス
そもそも論になりますが、Pythonのクラス定義の際には
class ExampleClass(ParentClass):
A = 1
...
としますが、これはどのような処理がなされているのでしょうか? 実はこのとき行われているのは以下のコードと等価です。
X = type('ExampleClass', (ParentClass), {'A':1})
このようにしてtypeクラスのインスタンスを作成することになるのですが、メタクラスを指定するとtypeの代わりにメタクラスのインスタンスを作成することになります。当然、メタクラスの__new__(),__init__()が実行されることになります。 今回のModelクラスのメタクラスであるContextMetaクラスは__new__()でModelクラスの__enter__()と__exit__()を作成しています。以下が実際のContextMetaクラスの__new__(),__init__()です。 以後コードを貼るときは本質的に理解に不要なコメントやエラーハンドリング等は省略します。
model.py/ContextMeta
class ContextMeta(type):
"""省略"""
def __new__(cls, name, bases, dct, **kwargs): # pylint: disable=unused-argument
"""Add __enter__ and __exit__ methods to the class."""
def __enter__(self):
self.__class__.context_class.get_contexts().append(self)
# self._pytensor_config is set in Model.__new__
self._config_context = None
if hasattr(self, "_pytensor_config"):
self._config_context = pytensor.config.change_flags(**self._pytensor_config)
self._config_context.__enter__()
return self
def __exit__(self, typ, value, traceback): # pylint: disable=unused-argument
self.__class__.context_class.get_contexts().pop()
# self._pytensor_config is set in Model.__new__
if self._config_context:
self._config_context.__exit__(typ, value, traceback)
dct[__enter__.__name__] = __enter__
dct[__exit__.__name__] = __exit__
def __init__(
cls, name, bases, nmspc, context_class: Optional[Type] = None, **kwargs
): # pylint: disable=unused-argument
"""Add ``__enter__`` and ``__exit__`` methods to the new class automatically."""
if context_class is not None:
cls._context_class = context_class
super().__init__(name, bases, nmspc)
そして、以下がModelクラスの__init__()までです。
model.py/Model
class Model(WithMemoization, metaclass=ContextMeta):
if TYPE_CHECKING:
def __enter__(self: "Model") -> "Model":
...
def __exit__(self: "Model", *exc: Any) -> bool:
...
def __new__(cls, *args, **kwargs):
# resolves the parent instance
instance = super().__new__(cls)
if kwargs.get("model") is not None:
instance._parent = kwargs.get("model")
else:
instance._parent = cls.get_context(error_if_none=False)
instance._pytensor_config = kwargs.get("pytensor_config", {})
return instance
"""省略"""
def __init__(
self,
name="",
coords=None,
check_bounds=True,
*,
pytensor_config=None,
model=None,
):
del pytensor_config, model # used in __new__
self.name = self._validate_name(name)
self.check_bounds = check_bounds
if self.parent is not None:
self.named_vars = treedict(parent=self.parent.named_vars)
"""省略"""
else:
self.named_vars = treedict()
"""省略"""
self.add_coords(coords)
from pymc.printing import str_for_model
self.str_repr = types.MethodType(str_for_model, self)
self._repr_latex_ = types.MethodType(
functools.partial(str_for_model, formatting="latex"), self
)
実装の確認
これを踏まえてwith文のところまでの挙動を見てみましょう。 まずはModelクラスの定義時にContextMeta.__new__()が実行され、Model.__enter__()とModel.__exit__()が定義されます。次にContextMeta.__init__()が実行されます。
with文実行時にModelクラスのインスタンスが作成されます。この際にはあまり変なことは起きません。 その次に今作ったインスタンスの__enter__()メソッドが実行されます。ここでのポイントが同メソッドで行われる
self.__class__.context_class.get_contexts().append(self)
です。ここでModelクラスの「今対象にしている同クラスのリスト」の末尾に今作ったモデルインスタンスが追加されます。 こうなっているため後々with文内でModelインスタンスを意識することなくモデルを組み立てられるようになっています。
確率変数と分布クラスの管理
分布クラスの構造
次に
p = pm.Beta("p", alpha=1, beta=1)
を見ていきましょう。二項分布のパラメータの事前分布としてベータ分布を与えるところです。 プログラム的にはベータ分布クラスのインスタンスを作っているので、どのようなことが行われているかをみていきます。
ベータ分布クラス
まずベータ分布に至るまでの継承とメタクラスの関係を見ていくと、 ベータ分布クラス→台が[0,1]の連続分布クラス→連続分布クラス→分布クラス→(メタクラス)分布メタクラス という関係になっています((子)→(親)の関係性)。
それらのうち、内容があるのはベータ分布クラス、分布クラス、分布メタクラスなので前2つを見ていきます。分布メタクラスの内容は省略します。
ベータ分布のコードが以下です。
distributions/continuous.py/Beta
class Beta(UnitContinuous):
"""省略"""
rv_op = pytensor.tensor.random.beta
@classmethod
def dist(cls, alpha=None, beta=None, mu=None, sigma=None, nu=None, *args, **kwargs):
alpha, beta = cls.get_alpha_beta(alpha, beta, mu, sigma, nu)
alpha = at.as_tensor_variable(floatX(alpha))
beta = at.as_tensor_variable(floatX(beta))
return super().dist([alpha, beta], **kwargs)
def moment(rv, size, alpha, beta):
"""省略"""
return mean
@classmethod
def get_alpha_beta(self, alpha=None, beta=None, mu=None, sigma=None, nu=None):
"""省略"""
return alpha, beta
def logp(value, alpha, beta):
"""省略"""
return check_parameters(
res,
alpha > 0,
beta > 0,
msg="alpha > 0, beta > 0",
)
def logcdf(value, alpha, beta):
"""省略"""
return check_parameters(
logcdf,
alpha > 0,
beta > 0,
msg="alpha > 0, beta > 0",
)
平均、パラメータ、対数密度、累積分布関数の対数値を得るためのメソッドが定義されています。__new__()は後で扱う分布クラスのものを使います。 dist()メソッドも後で扱いますが、これは__new__()実行時に呼び出されます。ベータ分布に従うPyTensorの確率変数クラスにパラメータを与えてインスタンス(rv_out)を得るためのメソッドです。ということで次に分布クラスを見ていきます。
分布クラス
分布クラスには個別の分布のインスタンスを生成するときの__new__()メソッドと、個別の分布クラスのdist()メソッドを内部で呼び出すdist()メソッドが実装されています。__new__()メソッドがdist()メソッドを呼び出すことで確率変数インスタンスrv_outを作り、その後Model.register_rv()メソッドで先程作ったModelインスタンスに登録します。このとき確率変数に付けた名前や対数尤度を計算する時の変形等も登録します。 また、作った確率変数が観測値を持つか否かで処理を変えます。今回は直接観測される値では無いので先程のModelインスタンスのfree_RVsに登録されます。 分布クラスを見て実際にそうなっていることを確認しましょう。
distributions/distributions.py/Distribution
class Distribution(metaclass=DistributionMeta):
rv_op: [RandomVariable, SymbolicRandomVariable] = None
rv_type: MetaType = None
def __new__(
cls,
name: str,
*args,
rng=None,
dims: Optional[Dims] = None,
initval=None,
observed=None,
total_size=None,
transform=UNSET,
**kwargs,
) -> TensorVariable:
try:
from pymc.model import Model
model = Model.get_context()
except TypeError:
"""省略"""
"""省略"""
rv_out = cls.dist(*args, **kwargs)
rv_out = model.register_rv(
rv_out,
name,
observed,
total_size,
dims=dims,
transform=transform,
initval=initval,
)
# add in pretty-printing support
rv_out.str_repr = types.MethodType(str_for_dist, rv_out)
rv_out._repr_latex_ = types.MethodType(
functools.partial(str_for_dist, formatting="latex"), rv_out
)
rv_out.logp = _make_nice_attr_error("rv.logp(x)", "pm.logp(rv, x)")
rv_out.logcdf = _make_nice_attr_error("rv.logcdf(x)", "pm.logcdf(rv, x)")
rv_out.random = _make_nice_attr_error("rv.random()", "pm.draw(rv)")
return rv_out
@classmethod
def dist(
cls,
dist_params,
*,
shape: Optional[Shape] = None,
**kwargs,
) -> TensorVariable:
"""省略"""
rv_out = cls.rv_op(*dist_params, size=create_size, **kwargs)
rv_out.logp = _make_nice_attr_error("rv.logp(x)", "pm.logp(rv, x)")
rv_out.logcdf = _make_nice_attr_error("rv.logcdf(x)", "pm.logcdf(rv, x)")
rv_out.random = _make_nice_attr_error("rv.random()", "pm.draw(rv)")
_add_future_warning_tag(rv_out)
return rv_out
確かに、PyTensorの確率分布クラスにパラメータを与えてインスタンスを作りそれをモデルに登録する、というプロセスはこのような方式であればどの確率変数でも同じなのでこのような構成になるのは納得できます。 では次にモデルクラスのregister_rv()メソッドをみてみましょう、といってもモデルにいろいろ登録するだけですが。
model.py/Model
class Model(WithMemoization, metaclass=ContextMeta):
"""省略"""
def register_rv(
self, rv_var, name, observed=None, total_size=None, dims=None, transform=UNSET, initval=None
):
name = self.name_for(name)
rv_var.name = name
_add_future_warning_tag(rv_var)
"""省略"""
if observed is None:
if total_size is not None:
raise ValueError("total_size can only be passed to observed RVs")
self.free_RVs.append(rv_var)
self.create_value_var(rv_var, transform)
self.add_named_variable(rv_var, dims)
self.set_initval(rv_var, initval)
else:
"""省略"""
rv_var = self.make_obs_var(rv_var, observed, dims, transform, total_size)
return rv_var
観測された確率変数
次に追うコードは以下です。
obs = pm.Binomial("obs", p=p, n=trials,
observed=successes,
)
先程事前分布にベータ分布をおいたパラメータを使った二項分布です。先程と同様に二項分布に至るまでの継承とメタクラスを見ていくと、 二項分布クラス→離散分布クラス→分布クラス→(メタクラス)分布メタクラス となっていて、構成はもちろんほぼ同じですし実行されることも概ね同じです。異なるところはインスタンス生成時のModel.register_rv()メソッド内の挙動です。引数observedに観測データを渡したことで変化し、Model.make_obs_var()メソッドが実行されます。さらにその中でModel.create_value_var()が実行され、モデルに登録されます。 Model.make_obs_var()メソッドを見てみましょう。
model.py/Model
class Model(WithMemoization, metaclass=ContextMeta):
"""省略"""
def make_obs_var(
self,
rv_var: TensorVariable,
data: np.ndarray,
dims,
transform: Union[Any, None],
total_size: Union[int, None],
) -> TensorVariable:
name = rv_var.name
data = convert_observed_data(data).astype(rv_var.dtype)
if data.ndim != rv_var.ndim:
raise ShapeError(
"Dimensionality of data and RV don't match.", actual=data.ndim, expected=rv_var.ndim
)
if pytensor.config.compute_test_value != "off":
"""省略"""
mask = getattr(data, "mask", None)
if mask is not None:
"""省略"""
else:
if sps.issparse(data):
data = sparse.basic.as_sparse(data, name=name)
else:
data = at.as_tensor_variable(data, name=name)
if total_size:
from pymc.variational.minibatch_rv import create_minibatch_rv
rv_var = create_minibatch_rv(rv_var, total_size)
rv_var.name = name
rv_var.tag.observations = data
self.create_value_var(rv_var, transform=None, value_var=data)
self.add_named_variable(rv_var, dims)
self.observed_RVs.append(rv_var)
return rv_var
make_obs_var()の最後の処理は、observedがNoneのときのModel.register_rv()の最後の処理とほとんど同じです。極論すると異なる部分はrv_var.tag.observations = dataを与えているかどうかのようです。
サンプリング
最後の一行、サンプリングの部分です。
idata = pm.sample()
サンプリング概観
最近のデータ分析環境であればまず間違いなくマルチコアCPUが使えるので、自動的に並列サンプリングが実行されます。 今回のコードを実行するとpymcで実装されたNUTSサンプリングが並列で走るので、そのケースがたどる部分を見ていこうと思います。 まずはsample()関数を見てみましょう。
sampling/mcmc.py/sample
def sample(
draws: int = 1000,
*,
tune: int = 1000,
chains: Optional[int] = None,
cores: Optional[int] = None,
random_seed: RandomState = None,
progressbar: bool = True,
step=None,
nuts_sampler: str = "pymc",
initvals: Optional[Union[StartDict, Sequence[Optional[StartDict]]]] = None,
init: str = "auto",
jitter_max_retries: int = 10,
n_init: int = 200_000,
trace: Optional[TraceOrBackend] = None,
discard_tuned_samples: bool = True,
compute_convergence_checks: bool = True,
keep_warning_stat: bool = False,
return_inferencedata: bool = True,
idata_kwargs: Optional[Dict[str, Any]] = None,
callback=None,
mp_ctx=None,
model: Optional[Model] = None,
**kwargs,
) -> Union[InferenceData, MultiTrace]:
"""省略"""
model = modelcontext(model)
if not model.free_RVs:
"""省略"""
if cores is None:
cores = min(4, _cpu_count())
if chains is None:
chains = max(2, cores)
"""省略"""
step = assign_step_methods(model, step, methods=pm.STEP_METHODS, step_kwargs=kwargs)
if nuts_sampler != "pymc":
"""省略"""
# Create trace backends for each chain
run, traces = init_traces(
backend=trace,
chains=chains,
expected_length=draws + tune,
step=step,
initial_point=ip,
model=model,
)
sample_args = {
"draws": draws + tune, # FIXME: Why is tune added to draws?
"step": step,
"start": initial_points,
"traces": traces,
"chains": chains,
"tune": tune,
"progressbar": progressbar,
"model": model,
"cores": cores,
"callback": callback,
"discard_tuned_samples": discard_tuned_samples,
}
parallel_args = {
"mp_ctx": mp_ctx,
}
sample_args.update(kwargs)
"""省略"""
if parallel:
_log.info(f"Multiprocess sampling ({chains} chains in {cores} jobs)")
_print_step_hierarchy(step)
try:
_mp_sample(**sample_args, **parallel_args)
except pickle.PickleError:
"""省略"""
if not parallel:
"""省略"""
return _sample_return(
run=run,
traces=traces,
tune=tune,
t_sampling=t_sampling,
discard_tuned_samples=discard_tuned_samples,
compute_convergence_checks=compute_convergence_checks,
return_inferencedata=return_inferencedata,
keep_warning_stat=keep_warning_stat,
idata_kwargs=idata_kwargs or {},
model=model,
)
コードからわかるように、ここではassign_step_methods()メソッドを使ってstepすなわちサンプリング手法を決定し、そのあと_mp_sample()に引数を入れて実行しています。実はassign_step_methods()はサンプリング手法を決定するだけの関数ではなく、各サンプリング手法クラスのインスタンスを作って返します。 ということで次はassign_step_methods()の中身を見ていきましょう。
サンプリング手法選定
この関数では、パラメータごとに各サンプリング手法(現在候補はNUTS, HMC, Metropolis,BinaryMetropolis, BinaryGibbsMetropolis, Slice, CategoricalGibbsMetropolis)から最適なものを選びます。各サンプリング手法はクラスとして存在しており、どの手法も.competence()という相性を測るメソッドが実装されています。このメソッドは0-3の点数を返し、大きい方がより良いとされています。NUTSを例に取ると、変数が対数尤度に対して勾配を持つ場合2点を返すようになっています。現状3点を返す手法はかなり限られているので、通常連続変数で勾配があればNUTSが選ばれるようです。
余談ですが3点を返すケースはとても少ないようです。例えば、ベルヌーイ分布に従うような確率変数に対しては、BinaryGibbsMetropolisサンプリングが理想的と判定されます。この辺なにか研究があるのでしょうか?気になるところではあります。
最後にinstantiate_steppersを実行してその返り値を返します。関数名の通り各サンプリング手法クラスのインスタンスを作り、それらを二個目の引数のリスト(steps)に足したものを返します。
sampling/mcmc.py/assign_step_methods
def assign_step_methods(model, step=None, methods=None, step_kwargs=None):
steps = []
assigned_vars = set()
if methods is None:
methods = pm.STEP_METHODS
if step is not None:
"""省略"""
# Use competence classmethods to select step methods for remaining
# variables
selected_steps = defaultdict(list)
model_logp = model.logp()
for var in model.value_vars:
if var not in assigned_vars:
# determine if a gradient can be computed
has_gradient = var.dtype not in discrete_types
if has_gradient:
try:
tg.grad(model_logp, var)
except (NotImplementedError, tg.NullTypeGradError):
has_gradient = False
# select the best method
rv_var = model.values_to_rvs[var]
selected = max(
methods,
key=lambda method, var=rv_var, has_gradient=has_gradient: method._competence(
var, has_gradient
),
)
selected_steps[selected].append(var)
return instantiate_steppers(model, steps, selected_steps, step_kwargs)
並列サンプリング
次にサンプリングの本丸、_mp_sample()メソッドをみていきます。 やっていることはParallelSamplerインスタンスを作ったあとにそこからイテレーション結果を引き出してtraceに格納しているだけです。
sampling/mcmc.py/_mp_sample
def _mp_sample(
*,
draws: int,
tune: int,
step,
chains: int,
cores: int,
random_seed: Sequence[RandomSeed],
start: Sequence[PointType],
progressbar: bool = True,
traces: Sequence[IBaseTrace],
model: Optional[Model] = None,
callback: Optional[SamplingIteratorCallback] = None,
mp_ctx=None,
**kwargs,
) -> None:
"""省略"""
import pymc.sampling.parallel as ps
# We did draws += tune in pm.sample
draws -= tune
sampler = ps.ParallelSampler(
draws=draws,
tune=tune,
chains=chains,
cores=cores,
seeds=random_seed,
start_points=start,
step_method=step,
progressbar=progressbar,
mp_ctx=mp_ctx,
)
try:
try:
with sampler:
for draw in sampler:
strace = traces[draw.chain]
strace.record(draw.point, draw.stats)
log_warning_stats(draw.stats)
if draw.is_last:
strace.close()
if callback is not None:
callback(trace=strace, draw=draw)
except ps.ParallelSamplingError as error:
"""省略"""
except KeyboardInterrupt:
pass
finally:
for strace in traces:
strace.close()
ここ自体はそんなに難しくありませんね。 では次にParallelSampelerクラスのインスタンス作成からイテレーション実行までを見ていきます。
初期化メソッドでまず並列処理の開始方式を決めています。mp_ctx関連の部分です。OSが変わってもきちんと動くようにうまくやっているという認識で大丈夫です。 次にチェーン数だけProcessAdapterインスタンスとそれに伴う子プロセスを作りリスト化した後、それら全てをself._inactiveというまだ動いていないプロセスに登録します。
イテレーション実行時には、ParallelSamplerの__iter__()メソッドが呼び出されます。__iter__()メソッドが呼び出されたときの一般的な挙動を詳細に述べると長くなるのでざっくりした説明が次のようになります。
for i in hoge: を実行するとhoge.__iter__()が実行され、最初のyieldのところまで実行して止まり、iにyeild以後を代入して返します。そしてループが回ってiの値を更新する際には、先程のyieldのところから再開し、次のyieldのところまで実行しiにそれを代入して返す、という挙動になります。
今回のコードの場合だと、最初にParallelSamplerの._make_active()メソッドを実行して各プロセスに対しproc.start()メソッドとproc.write_next()メソッドを実行します。proc.start()メソッドで'start'メッセージを受け取った子プロセス達は各々サンプリングを始め、'write_next'メッセージ=パイプに結果を書き込んで良いよ、のメッセージを受取るまで結果を保持して待ちます。親プロセス側でProcessAdapter.recv_draw()が実行されると、最も早く結果が用意できたプロセスのパイプからサンプリング結果を取得します。その後proc.write_next()メソッドを呼ぶことで、結果を返した子プロセスに対し次のステップのサンプリング結果を計算して良いぞとパイプ越しに命令することになります。
sampling/parallel.py/ParallelSampler
class ParallelSampler:
def __init__(
self,
*,
draws: int,
tune: int,
chains: int,
cores: int,
seeds: Sequence["RandomSeed"],
start_points: Sequence[Dict[str, np.ndarray]],
step_method,
progressbar: bool = True,
mp_ctx=None,
):
""" 省略"""
if mp_ctx is None or isinstance(mp_ctx, str):
""" 省略"""
mp_ctx = multiprocessing.get_context(mp_ctx)
step_method_pickled = None
if mp_ctx.get_start_method() != "fork":
step_method_pickled = cloudpickle.dumps(step_method, protocol=-1)
self._samplers = [
ProcessAdapter(
draws,
tune,
step_method,
step_method_pickled,
chain,
seed,
start,
mp_ctx,
)
for chain, seed, start in zip(range(chains), seeds, start_points)
]
self._inactive = self._samplers.copy()
self._finished: List[ProcessAdapter] = []
self._active: List[ProcessAdapter] = []
self._max_active = cores
""" 省略"""
def _make_active(self):
while self._inactive and len(self._active) < self._max_active:
proc = self._inactive.pop(0)
proc.start()
proc.write_next()
self._active.append(proc)
def __iter__(self):
if not self._in_context:
raise ValueError("Use ParallelSampler as context manager.")
self._make_active()
if self._active and self._progress:
self._progress.update(self._total_draws)
while self._active:
draw = ProcessAdapter.recv_draw(self._active)
proc, is_last, draw, tuning, stats = draw
self._total_draws += 1
""" 省略"""
# Already called for new proc in _make_active
if not is_last:
proc.write_next()
yield Draw(proc.chain, is_last, draw, tuning, stats, point)
""" 省略"""
実際に親子間でやりとりするコードを見てみましょう。主にサンプリングのところだけを抜き出しています。終了処理も省略しています。 親プロセス側で、子プロセスと直接やりとりをするのが以下のProcessAdapterクラスです。
sampling/paralell.py/ProcessAdapter
class ProcessAdapter:
"""Control a Chain process from the main thread."""
def __init__(
self,
draws: int,
tune: int,
step_method,
step_method_pickled,
chain: int,
seed,
start: Dict[str, np.ndarray],
mp_ctx,
):
self.chain = chain
process_name = "worker_chain_%s" % chain
self._msg_pipe, remote_conn = multiprocessing.Pipe()
self._shared_point = {}
self._point = {}
for name, shape, dtype in DictToArrayBijection.map(start).point_map_info:
""" 省略"""
self._readable = True
self._num_samples = 0
if step_method_pickled is not None:
step_method_send = step_method_pickled
else:
if mp_ctx.get_start_method() == "spawn":
raise ValueError(
"please provide a pre-pickled step method when multiprocessing start method is 'spawn'"
)
step_method_send = step_method
self._process = mp_ctx.Process(
daemon=True,
name=process_name,
target=_run_process,
args=(
process_name,
remote_conn,
step_method_send,
step_method_pickled is not None,
self._shared_point,
draws,
tune,
seed,
),
)
self._process.start()
# Close the remote pipe, so that we get notified if the other
# end is closed.
remote_conn.close()
"""省略"""
def _send(self, msg, *args):
try:
self._msg_pipe.send((msg, *args))
except Exception:
# try to receive an error message
message = None
try:
message = self._msg_pipe.recv()
except Exception:
pass
if message is not None and message[0] == "error":
old_error = message[1]
if old_error is not None:
error = ParallelSamplingError(
f"Chain {self.chain} failed with: {old_error}", self.chain
)
else:
error = RuntimeError(f"Chain {self.chain} failed.")
raise error from old_error
raise
def start(self):
self._send("start")
def write_next(self):
self._readable = False
self._send("write_next")
""" 省略"""
@staticmethod
def recv_draw(processes, timeout=3600):
if not processes:
raise ValueError("No processes.")
pipes = [proc._msg_pipe for proc in processes]
ready = multiprocessing.connection.wait(pipes)
if not ready:
raise multiprocessing.TimeoutError("No message from samplers.")
idxs = {id(proc._msg_pipe): proc for proc in processes}
proc = idxs[id(ready[0])]
msg = ready[0].recv()
if msg[0] == "error":
""" 省略"""
elif msg[0] == "writing_done":
proc._readable = True
proc._num_samples += 1
return (proc,) + msg[1:]
else:
raise ValueError("Sampler sent bad message.")
""" 省略"""
def _run_process(*args):
_Process(*args).run()
一方で、子プロセスとして動かすのが以下のProcessクラスです。 self.recv_msg()でパイプからメッセージを受け取る際には何か受け取れるまで待ち続けます。 これらを見ると大まかな挙動がわかるのではないかと思います。
sampling/parallel.py/_Process
class _Process:
def __init__(
self,
name: str,
msg_pipe,
step_method,
step_method_is_pickled,
shared_point,
draws: int,
tune: int,
seed,
):
self._msg_pipe = msg_pipe
self._step_method = step_method
self._step_method_is_pickled = step_method_is_pickled
self._shared_point = shared_point
self._seed = seed
self._at_seed = seed + 1
self._draws = draws
self._tune = tune
def _unpickle_step_method(self):
""" 省略"""
def run(self):
try:
self._unpickle_step_method()
self._point = self._make_numpy_refs()
self._start_loop()
except KeyboardInterrupt:
pass
except BaseException as e:
e = ExceptionWithTraceback(e, e.__traceback__)
self._msg_pipe.send(("error", e))
self._wait_for_abortion()
finally:
self._msg_pipe.close()
""" 省略"""
def _recv_msg(self):
return self._msg_pipe.recv()
def _start_loop(self):
np.random.seed(self._seed)
draw = 0
tuning = True
msg = self._recv_msg()
if msg[0] == "abort":
raise KeyboardInterrupt()
if msg[0] != "start":
raise ValueError("Unexpected msg " + msg[0])
while True:
if draw == self._tune:
self._step_method.stop_tuning()
tuning = False
if draw < self._draws + self._tune:
try:
point, stats = self._step_method.step(self._point)
except SamplingError as e:
e = ExceptionWithTraceback(e, e.__traceback__)
self._msg_pipe.send(("error", e))
else:
return
msg = self._recv_msg()
if msg[0] == "abort":
raise KeyboardInterrupt()
elif msg[0] == "write_next":
self._write_point(point)
is_last = draw + 1 == self._draws + self._tune
self._msg_pipe.send(("writing_done", is_last, draw, tuning, stats))
draw += 1
else:
raise ValueError("Unknown message " + msg[0])
NUTS実装
これまでで概ね実装は追い終わり、PyMCが何を担当しているかがわかってきました。MCMCサンプリングを通じて、PyMCは確率変数とそれらの関係性をモデルとして管理することと、並列サンプリングをしていました。逆に、確率分布そのもの実装や対数尤度の計算はPyTensorにまかせていることもわかってきました。 若干オプショナルになりますが、PyMCによるNUTSの実装も読んでみることにします。オプショナルなのは単純な理由で、PyMCで実装されているNUTSはJAXを使ったNUTSよりも遅いため使う必要性があまり無いからです。実際、公式も最新バージョン(5.1.2)ではsample()関数の引数からNUTSサンプラーにnutpieを選ぶのが最速とアナウンスしています。ですが、今回のコードだとサンプラーを指定していないのでPyMCのものが動くのと、そしてNUTSはどう実装されているのかを確認したいという個人的欲求から見ていくことにします。NUTSやHMCサンプリングについては[6]で確認してください。
これから見ていくところは、Processクラスのstart_loop()メソッド内にある point, stats = self.step_method.step(self.point) を行ったときに呼び出される各サンプリングクラスの.step()とそれに関連する部分です。
まずはNUTSクラスの継承関係を見ていくと、 NUTS→BaseHMC→GradientSharedStep→ArrayStepShared→BlockedStep という関係になっています。 NUTS.step()を実行したときに実際に実行されるのはArrayStepSharedから継承した.step()メソッドですが、さらに詳細に見るとBaseHMC.astep()メソッドが呼ばれています。 その中では初速の生成、対数尤度からのエネルギーの計算、1ステップ分のサンプリング、(ステップサイズを自動更新するなら)ステップサイズの更新、発散状況の警告作成等を行います。統計に近いのでやっていることがわかりやすいです。 コードは以下のようになっています。
step_methods/hmc/base_hmc.py/BaseHMC
class BaseHMC(GradientSharedStep):
"""省略"""
def astep(self, q0: RaveledVars) -> tuple[RaveledVars, StatsType]:
"""Perform a single HMC iteration."""
perf_start = time.perf_counter()
process_start = time.process_time()
p0 = self.potential.random()
p0 = RaveledVars(p0, q0.point_map_info)
start = self.integrator.compute_state(q0, p0)
warning: SamplerWarning | None = None
if not np.isfinite(start.energy):
"""省略"""
raise SamplingError(f"Bad initial energy: {warning}")
adapt_step = self.tune and self.adapt_step_size
step_size = self.step_adapt.current(adapt_step)
self.step_size = step_size
if self._step_rand is not None:
step_size = self._step_rand(step_size)
hmc_step = self._hamiltonian_step(start, p0.data, step_size)
perf_end = time.perf_counter()
process_end = time.process_time()
self.step_adapt.update(hmc_step.accept_stat, adapt_step)
self.potential.update(hmc_step.end.q, hmc_step.end.q_grad, self.tune)
if hmc_step.divergence_info:
info = hmc_step.divergence_info
point = None
point_dest = None
info_store = None
if self.tune:
kind = WarningType.TUNING_DIVERGENCE
else:
kind = WarningType.DIVERGENCE
self._num_divs_sample += 1
# We don't want to fill up all memory with divergence info
if self._num_divs_sample < 100 and info.state is not None:
point = DictToArrayBijection.rmap(info.state.q)
if self._num_divs_sample < 100 and info.state_div is not None:
point_dest = DictToArrayBijection.rmap(info.state_div.q)
if self._num_divs_sample < 100:
info_store = info
warning = SamplerWarning(
kind,
info.message,
"debug",
self.iter_count,
info.exec_info,
divergence_point_source=point,
divergence_point_dest=point_dest,
divergence_info=info_store,
)
self.iter_count += 1
stats: dict[str, Any] = {
"tune": self.tune,
"diverging": bool(hmc_step.divergence_info),
"perf_counter_diff": perf_end - perf_start,
"process_time_diff": process_end - process_start,
"perf_counter_start": perf_start,
"warning": warning,
}
stats.update(hmc_step.stats)
stats.update(self.step_adapt.stats())
stats.update(self.potential.stats())
return hmc_step.end.q, [stats]
1ステップ分のサンプリングだけはNUTS特有ですが、ほかの部分はHMC系?のサンプリングで共通なのでこのようにbaseHMCクラスのような実装になっています。
NUTSのサンプリングと言えば、前後のどちらかに進むかをランダムに決めて1ステップそちらに時間積分で進み、また進む方向を決めて2ステップ進み、次は4ステップ…を繰り返しそれを木としてくっつけていく手法です。1サイクルごとに積分した軌跡がパラメータ空間中でUターンしていないことを確認していくことが名前の由来です。こちらの実装も見てみましょう。論文中の擬似コードをそのまま書き写したといった感じでこちらもわかりやすくなっています。
step_methods/hmc/nuts.py NUTS
class NUTS(BaseHMC):
"""省略"""
name = "nuts"
default_blocked = True
stats_dtypes_shapes = {
"depth": (np.int64, []),
"""省略"""
}
def __init__(self, vars=None, max_treedepth=10, early_max_treedepth=8, **kwargs):
"""省略"""
super().__init__(vars, **kwargs)
self.max_treedepth = max_treedepth
self.early_max_treedepth = early_max_treedepth
self._reached_max_treedepth = 0
def _hamiltonian_step(self, start, p0, step_size):
if self.tune and self.iter_count < 200:
max_treedepth = self.early_max_treedepth
else:
max_treedepth = self.max_treedepth
tree = _Tree(len(p0), self.integrator, start, step_size, self.Emax)
reached_max_treedepth = False
for _ in range(max_treedepth):
direction = logbern(np.log(0.5)) * 2 - 1
divergence_info, turning = tree.extend(direction)
if divergence_info or turning:
break
else:
reached_max_treedepth = not self.tune
stats = tree.stats()
accept_stat = stats["mean_tree_accept"]
stats["reached_max_treedepth"] = reached_max_treedepth
return HMCStepData(tree.proposal, accept_stat, divergence_info, stats)
そして以下がサンプリング経路を保持するTreeクラスです。 build_subtree()メソッドは一回実行されるごとに二回再帰的に実行されるため、本体のツリーに追加されるサブツリーがステップごとに倍々になっていきます。
step_methods/hmc/nuts.py _Tree
class _Tree:
def __init__(
self,
ndim: int,
integrator: integration.CpuLeapfrogIntegrator,
start: State,
step_size: float,
Emax: float,
):
"""省略"""
def extend(self, direction):
"""省略"""
if direction > 0:
tree, diverging, turning = self._build_subtree(
self.right, self.depth, floatX(np.asarray(self.step_size))
)
leftmost_begin, leftmost_end = self.left, self.right
rightmost_begin, rightmost_end = tree.left, tree.right
leftmost_p_sum = self.p_sum.copy()
rightmost_p_sum = tree.p_sum
self.right = tree.right
else:
"""省略"""
self.depth += 1
if diverging or turning:
return diverging, turning
size1, size2 = self.log_size, tree.log_size
if logbern(size2 - size1):
self.proposal = tree.proposal
self.log_size = np.logaddexp(self.log_size, tree.log_size)
self.p_sum[:] += tree.p_sum
# Additional turning check only when tree depth > 0 to avoid redundant work
if self.depth > 0:
left, right = self.left, self.right
p_sum = self.p_sum
turning = (p_sum.dot(left.v) <= 0) or (p_sum.dot(right.v) <= 0)
p_sum1 = leftmost_p_sum + rightmost_begin.p.data
turning1 = (p_sum1.dot(leftmost_begin.v) <= 0) or (p_sum1.dot(rightmost_begin.v) <= 0)
p_sum2 = leftmost_end.p.data + rightmost_p_sum
turning2 = (p_sum2.dot(leftmost_end.v) <= 0) or (p_sum2.dot(rightmost_end.v) <= 0)
turning = turning | turning1 | turning2
return diverging, turning
def _single_step(self, left: State, epsilon: float):
"""Perform a leapfrog step and handle error cases."""
"""省略"""
return tree, divergence_info, False
def _build_subtree(self, left, depth, epsilon):
if depth == 0:
return self._single_step(left, epsilon)
tree1, diverging, turning = self._build_subtree(left, depth - 1, epsilon)
if diverging or turning:
return tree1, diverging, turning
tree2, diverging, turning = self._build_subtree(tree1.right, depth - 1, epsilon)
left, right = tree1.left, tree2.right
if not (diverging or turning):
p_sum = tree1.p_sum + tree2.p_sum
turning = (p_sum.dot(left.v) <= 0) or (p_sum.dot(right.v) <= 0)
# Additional U turn check only when depth > 1 to avoid redundant work.
if depth - 1 > 0:
p_sum1 = tree1.p_sum + tree2.left.p.data
turning1 = (p_sum1.dot(tree1.left.v) <= 0) or (p_sum1.dot(tree2.left.v) <= 0)
p_sum2 = tree1.right.p.data + tree2.p_sum
turning2 = (p_sum2.dot(tree1.right.v) <= 0) or (p_sum2.dot(tree2.right.v) <= 0)
turning = turning | turning1 | turning2
log_size = np.logaddexp(tree1.log_size, tree2.log_size)
if logbern(tree2.log_size - log_size):
proposal = tree2.proposal
else:
proposal = tree1.proposal
else:
p_sum = tree1.p_sum
log_size = tree1.log_size
proposal = tree1.proposal
tree = Subtree(left, right, p_sum, proposal, log_size)
return tree, diverging, turning
def stats(self):
self.mean_tree_accept = np.exp(self.log_accept_sum) / self.n_proposals
return {
"""省略"""
}
まとめ
モデル作成からサンプリングまでの挙動を見ることで、PyMCが実際に何をしているのかを明らかにしました。 PyMCが担当しているパートはおおまかに言うと
- PyTensorの確率変数をインスタンス化
- モデルとして確率変数の関係を保持
- サンプリングの並列実行
- サンプリング手法の実装
でした。理解が深まる一助になれば幸いです。
[1]: https://pymc-devs.medium.com/theano-tensorflow-and-the-future-of-pymc-6c9987bb19d5
[2]: https://pymc-devs.medium.com/the-future-of-pymc3-or-theano-is-dead-long-live-theano-d8005f8a0e9b
[3]: https://www.pymc.io/about/history.html
[4]: https://www.pymc.io/blog/pytensor_announcement.html#pytensor_announcement
[5]: https://docs.python.org/ja/3/reference/compound_stmts.html#with
[6]: https://arxiv.org/pdf/1111.4246.pdf